| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | |||
| 5 | 6 | 7 | 8 | 9 | 10 | 11 |
| 12 | 13 | 14 | 15 | 16 | 17 | 18 |
| 19 | 20 | 21 | 22 | 23 | 24 | 25 |
| 26 | 27 | 28 | 29 | 30 | 31 |
Tags
- keras
- 판다스
- DANAWA
- 자바스크립트
- data analyze
- data
- webcrawling
- Method
- matplotlib
- Project
- angular
- algorithm
- tensorflow
- python
- 프로젝트
- Scrum
- 다나와
- Crawling
- javascript
- 애자일
- ECS
- opencv
- analyzing
- Agile
- adaptive life cycle
- pandas
- visualizing
- 크롤링
- TypeScript
- AWS
Archives
- Today
- Total
LiJell's 성장기
군집화 (Clustering) 본문
반응형
- 비지도 학습
- 데이터의 성질로 부터 최적으로 분할하고 레이블을 구함
- 활용 분야
- 시장 분할
- 지도에서 가까운 지점을 병함
- 영상 압축
- 자료에 새 레이블 부여
- 이상행동 감지
- 방법론
- K-means clustering, DBSCAN, ect
K- 평균 군집화
- 레이블이 없는 다차원 데이터 세트 내에 사전 정의된 군집의 개수를 찾아내는 방법
- 최적의 군집화
- ''군집의 중앙' 은 해당 군집에 속하는 모든 점의 산술 평균
- 각 점은 다른 군집의 중앙보다 자신이 속한 군집의 중앙에 더 가깝다.
%matplotlib inline
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
import numpy as np# 4개 영역의 2차원 자료의 생성
from sklearn.datasets import make_blobs
# y_ture 는 cluster의 num. X는 dot의 좌표값. 따라서 아래에서는 사실 필요없음
X, y_true = make_blobs(n_samples=300, centers=4,
cluster_std = 0.60, random_state = 0)
plt.scatter(X[:,0], X[:,1], s=50)

# k-means clustering
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters = 4)
y_kmeans = kmeans.fit_predict(X)
print(y_kmeans)
'''
[2 3 1 3 2 2 0 1 3 3 0 3 1 3 2 1 1 2 0 0 2 2 1 0 0 1 2 1 0 1 3 3 1 3 3 3 3
3 0 2 1 0 1 1 0 0 3 0 3 2 0 2 3 2 2 0 3 0 3 2 3 1 3 0 0 0 3 2 3 0 1 0 3 0
0 3 0 1 2 3 2 1 2 2 3 1 2 1 3 3 1 2 3 0 0 1 2 2 1 0 3 2 3 2 1 2 2 1 3 1 0
0 2 3 2 1 3 2 2 1 0 2 0 2 2 2 2 0 2 0 3 0 0 2 3 0 0 3 1 3 3 0 1 0 1 0 3 1
3 3 3 1 3 1 2 0 3 0 2 1 3 1 1 2 1 0 0 1 2 1 1 3 2 1 0 3 2 2 1 0 2 1 0 0 1
1 1 1 2 3 1 0 1 1 0 0 0 1 0 3 1 0 2 0 1 3 0 3 1 3 1 0 1 1 3 0 0 2 2 1 3 2
2 0 2 0 1 3 3 1 1 3 1 2 0 1 2 0 3 0 2 1 2 3 3 3 3 0 0 3 1 0 2 1 0 0 0 2 2
3 1 1 0 2 3 0 1 3 1 2 2 0 0 1 2 2 2 1 3 3 2 2 1 2 2 2 3 0 3 1 2 2 3 3 3 2
2 1 3 0]
'''# 그룹별로 색깔을 달리 표현하기, 군집 중앙표시
plt.scatter(X[:,0], X[:,1], c=y_kmeans, s=50, cmap = 'viridis')
centers = kmeans.cluster_centers_
plt.scatter(centers[:,0], centers[:,1], c='k', s=200, alpha=0.5)
K-means 군집화 알고리즘
- 기대값-최대화(E-M) 알고리즘② 수렴될 때까지 다음을 반복한다.
- E-단계(기댓값 단계): 점을 가장 가까운 군집 중심에 할당한다.
- M-단계(최대화 단계): 군집 중심을 평균값으로 설정한다. (군집에 속한 데이터의 산술 평균)
- ① 일부 군집 중심을 추측한다.(난수 초기값)
기대값-최대화(E-M) 알고리즘 관련 주의사항
- 최초의 군집 중심을 난수 초기값으로 정하기 때문에 최적화된 결과를 얻지 못하는 경우도 있다.
- 군집의 개수가 사전에 정해져야 한다.
- K-평균 군집은 선형 군집 경계로 한정된다. (치명적 단점)
- K-평균 균집은 표본 수가 많아지면 느려진다
- 알고리즘을 반복할 때마다 데이터세트의 모든 점에 접근해야 하므로
# 비선형 경계를 가지는 자료의 경우
from sklearn.datasets import make_moons
X, y = make_moons(200, noise=0.05, random_state = 0)
labels = KMeans(2, random_state = 0).fit_predict(X)
plt.scatter(X[:,0], X[:,1], c=labels, s=50, cmap='viridis')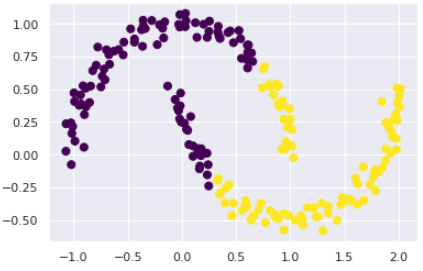
# 필기체 숫자 인식에 적용
from sklearn.datasets import load_digits
digits = load_digits()
digits.data.shape
'''
(1797, 64)
'''# k-meansa clustering
from sklearn.cluster import KMeans
kmeans = KMeans(n_clusters= 10, random_state= 0)
clusters = kmeans.fit_predict(digits.data)
kmeans.cluster_centers_.shape
'''
(10, 64)
'''# 64차원의 군집 10개
fig, ax = plt.subplots(2,5, figsize=(8,3)) # ax는 축
centers = kmeans.cluster_centers_.reshape(10,8,8)
for axi, center in zip(ax.flat, centers):
axi.set(xticks=[], yticks=[])
axi.imshow(center, interpolation = 'nearest', cmap=plt.cm.binary)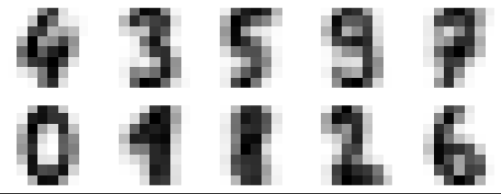
DBSCAN
- Density Based Spatial Clustering of Application with Noise
- 선형 군집 경계로 한정되는 K-평균 군집의 단점을 보완
특징
- 노이즈에 강한 군집 모델
- 밀도있게 연결되어 있는 데이터 집합을 동일한 클러스터로 결정함
- 일정한 밀도를 가지는 데이터 무리가 체인처럼 연결되어 있으면 거리의 개념과 관계없이 같은 클러스터로 판단함
- cluster의 개수가 정해져잇지 않음
DBSCAN 용어
X : 학습 데이터 전체 집합
𝜀 ∶ 밀도측정 반지름
𝑀𝑖𝑛𝑃𝑡𝑠 : 반지름 𝜀 이내에 있는 최소 데이터 개수
N(x) : 데이터 x의 반지름 𝜀 내에 있는 이웃 데이터(neighbor) 수
{x} : 데이터 x의 반지름 𝜀 내에 있는 이웃 데이터
- x is 𝑥𝑐𝑜𝑟𝑒 if N(x) ≥ 𝑀𝑖𝑛𝑃𝑡𝑠 ∀𝑥∈𝑋
- x is 𝑥𝑏𝑜𝑟𝑑𝑒𝑟 if x∈ {𝑥𝑐𝑜𝑟𝑒 } 이고 N(x) < 𝑀𝑖𝑛𝑃𝑡𝑠 ∀𝑥∈𝑋
- x is 𝑥𝑛𝑜𝑖𝑠𝑒 if x∉ {𝑥𝑐𝑜𝑟𝑒 } 이고 N x < 𝑀𝑖𝑛𝑃𝑡𝑠 ∀𝑥∈𝑋
DBSCAN 알고리즘
- 밀도 반지름 𝜀 반경 내 최소 데이터 개수(MinPts) 정의, C=0
- 모든 데이터 x ∈X에 대하여 다음을 수행2-2) 만약 N(x) < MinPts 이면 ② 2단계로 돌아가 다른 데이터로 다시 시작 (코어가 없으면 ① x는 코어 ③ x의 밀도 반지름에 속해있는 모든 점들에 대해 2단계 반복
- ② x가 아직 소속 클러스터가 없으면 C 할당
- 2-3) 만약 N(x) > MinPts 이면
- ① x는 Noise, C=C+1
- 2-1) x에 처음 방문하면 방문했다고 표시
- 군집화의 과정은 코어->코어->코어-> …->경계 방향
DBSCAN
from sklearn.datasets import make_moons
X, y = make_moons(200, noise=0.05, random_state = 0)from sklearn.cluster import DBSCAN
D_labels = DBSCAN(eps=0.3, min_samples= 15).fit_predict(X)
plt.scatter(X[:,0], X[:,1], c=D_labels, s=50, cmap='viridis')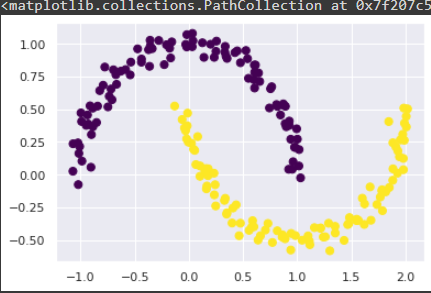
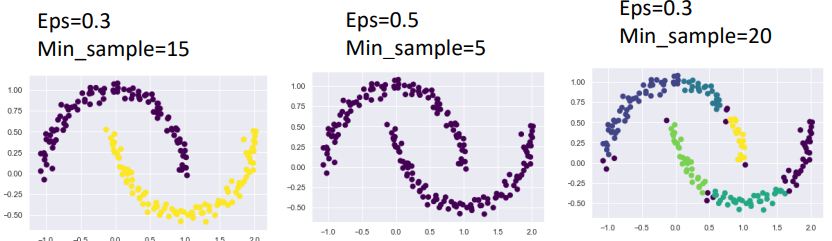
장점
- 도넛 모양이나 반달 모양의 데이터 세트에 대한 군집 가능

단점
- 밀도 반지름 및 최소 이웃 수가 문제의 특성에 따라 민감하게 작용함
비지도 학습: 주성분 분석 (PCA)
- PCA(Principal Component Analysis)
- 주성분 정보를 벡터와 길이로 분석
- 활용 분야
- 데이터의 주축(principal axes)의 목록을 구하고, 그 축을 사용해 데이터 세트를 설명
- 특징 추출
- 데이터의 분산 정보를 가장 많이 포함하는 주축으로 차원 축소
- ① 주성분 분석:
1. 주성분 분석
#PCA 를 위한 자료 준비
rng = np.random.RandomState(1)
X = np.dot(rng.rand(2,2), rng.randn(2, 200)).T
plt.scatter(X[:,0], X[:,1])
plt.axis('equal')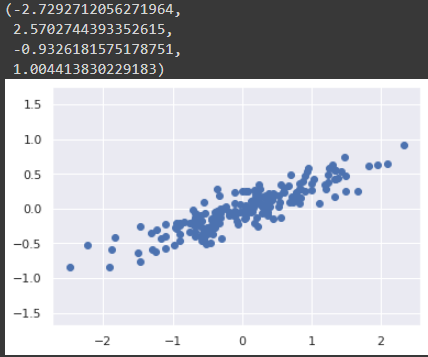
- 성분(component): 벡터의 방향
- 분산이 큰 순서로 PC 축이 차례로 생김. 예) component 1 = PC1 , component 2 = PC2
- PC 기준으로 축 변경하여 새로운 좌표가 생김
- 설명 분산(explained variance): 해당 벡터의 제곱 길이
from sklearn.decomposition import PCA
mypca = PCA(n_components = 2)
mypca.fit(X)print(mypca.components_)
'''
[[-0.94446029 -0.32862557] # component1 의 좌표
[-0.32862557 0.94446029]] # component 2 의 좌표
'''print(mypca.explained_variance_)
'''
[0.7625315 0.0184779] # component1까지의 거리, component2까지의 거리
'''- 이 두가지 기준으로 PC축이 생김
축 만들기 함수
def draw_vector(V0, V1, ax=None):
ax = ax or plt.gca() # gca()는 현재 Axes 객체를 반환한다.(get current axes)
arrowprops = dict(color = 'r',
arrowstyle='simple',
linewidth=2,
shrinkA=0,
shrinkB=0)
ax.annotate('', v1, v0, arrowprops=arrowprops)
# data plotting
plt.scatter(X[:,0], X[:,1], alpha=0.2)
for length, vector in zip(mypca.explained_variance_, mypca.components_):
v = vector * 3 * np.sqrt(length)
draw_vector(mypca.mean_, mypca.mean_ + v)
plt.axis('equal')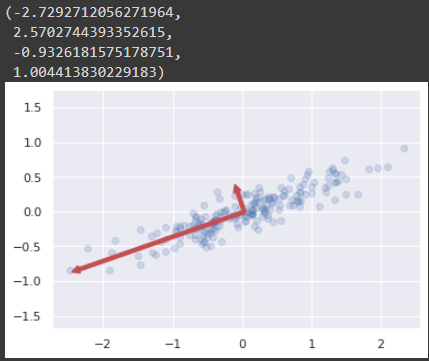
차원축소에 응용
- 가장 작은 주성분 중 하나를 삭제해 최대 데이터 분산을 보존하는 더 작은 차원으로 데이터를 사영함
- 가장 작은 주성분 = 분산이 제일 적은 PC 제외하여 차원을 축소함 = 오차를 최소화
dimpca = PCA(n_components=1)
dimpca.fit(X)
X_pca = dimpca.transform(X)
print('original shape: ', X.shape)
print('transformed shape: ', X_pca.shape)
'''
original shape: (200, 2)
transformed shape: (200, 1)
'''- 자료 출력
# 역변환
X_new = dimpca.inverse_transform(X_pca)
plt.scatter(X[:,0], X[:,1], alpha = 0.2)
plt.scatter(X_new[:,0], X_new[:,1], alpha = 0.8)
plt.axis('equal')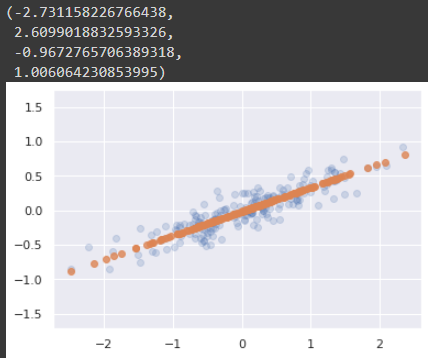
2. 특징 추출: 얼굴 특징 추출
# 고유 얼굴 성분 찾기
from sklearn.datasets import fetch_lfw_people
faces = fetch_lfw_people(min_faces_per_person=60)
print(faces.target_names)
print(faces.images.shape)
'''
['Ariel Sharon' 'Colin Powell' 'Donald Rumsfeld' 'George W Bush'
'Gerhard Schroeder' 'Hugo Chavez' 'Junichiro Koizumi' 'Tony Blair']
(1348, 62, 47)
'''from sklearn.decomposition import PCA
face_pca = PCA(150)
face_pca.fit(faces.data)
'''
PCA(n_components=150)
'''fig, axes = plt.subplots(3,8, figsize=(9,4),
subplot_kw = {'xticks': [], 'yticks': []},
gridspec_kw=dict(hspace=0.1, wspace=0.1))
for i, ax in enumerate(axes.flat):
ax.imshow(face_pca.components_[i].reshape(62,47), cmap='bone')
성능평가
예측 결과 평가 종류
- TP(True Positive): 실제 양성인데, 검사 결과 양성
- TN(True Negative): 실제는 음성인데, 검사 결과 음성
- FP(False Positive): 실제는 음성인데 검사결과는 양성(거짓 양성)
- FN(False Negative): 실제는 양성인데, 검사결과는 음성(거짓 음성)
| 실제 양성 | 실제 음성 | |
|---|---|---|
| 검사 양성 | TP | FP |
| 검사 음성 | FN | TN |
| 결과의 T or F \ 검사결과 | 양성 | 음성 |
|---|---|---|
| True | TP | TN |
| False | FP | FN |
성능
- 정확도(accuracy) = (𝑇𝑃+𝑇𝑁) / (𝑇𝑃+𝑇𝑁+𝐹𝑃+𝐹𝑁)
- 정밀도(precision) = 𝑇𝑃 / (𝑇𝑃+𝐹𝑃)
- 재현율(recall) = 𝑇𝑃 / (𝑇𝑃+𝐹𝑁)
- 민감도(Sensitivity) = 𝑇𝑃 / (𝑇𝑃+𝐹𝑃)
- 특이도(Specificity) = 𝑇𝑁 / (𝑇𝑁+𝐹𝑃)
반응형
Comments
